Utilisation des propriétés de la PCR pour calculer la quantité d'ADN amplifiée, ou bien la quantité d'ADN matrice soumis à amplification
La PCR peut être utilisée comme une approche quantitative lorsqu'on s'intéresse à la phase d'amplification exponentielle.
Méthode :
En effet, dans cette situation, la quantité d'ADN obtenue à l'issue de n cycles d'amplification Qn se détermine à partir de la quantité d'ADN obtenue à l'issue du cycle précédent Qn-1 de la façon suivante :
![]() (1)
(1)
où E est l'efficacité de la duplication de l'ADN.
Si l'efficacité de duplication est de 100% (E = 1), on a un doublement de la quantité d'ADN : Qn = 2 Qn-1. Si l'efficacité de duplication est plus faible, par exemple 90% (E = 0,9), la relation devient Qn = 1,9 Qn-1.
Par extension, on peut calculer la quantité d'ADN obtenue à l'issue de n cycles d'amplification d'ADN Qn à partir de la quantité initiale d'ADN Q0 de la façon suivante :
![]() (2)
(2)
Inversement, on peut calculer la quantité initiale d'ADN si on connaît la quantité finale :
![]() (3)
(3)
Fondamental :
On se trouve alors dans une logique quantitative, et on parle de PCR quantitative.
La formule :
|
est LA formule fondamentale à partir de laquelle tous les calculs quantitatifs peuvent être réalisés.
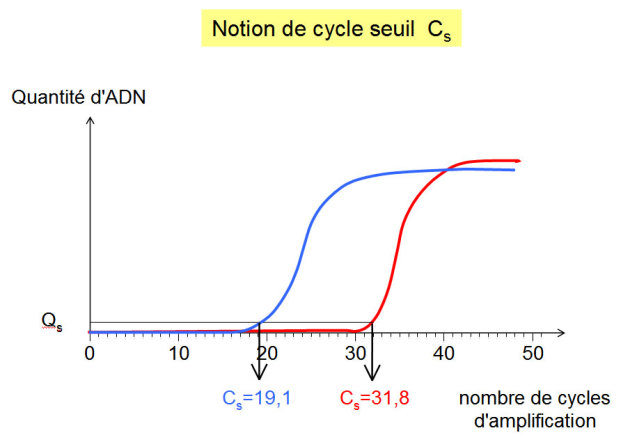
Notion de "cycle seuil" et utilisation pour l'analyse quantitave
Comme on l'a vu précédemment, pour déterminer la quantité initiale d'un ADN d'intérêt dans un échantillon en utilisant une approche de PCR quantitative, il faut analyser les échantillons lorsque le processus d'amplification est en phase exponentielle. Plus la mesure de la quantité d'ADN dans l'échantillon est précoce, plus l'expérimentateur a de chances que cette mesure ait lieu en phase exponentielle.
Par construction, les appareils de PCR quantitative utilisent une ‘valeur seuil' de fluorescence à partir de laquelle ils déterminent pour chaque échantillon le nombre de cycles d'amplification nécessaires pour atteindre ce seuil. La ‘valeur seuil' de fluorescence est environ 10 fois supérieure au bruit de fond de fluorescence. Elle reste cependant suffisamment faible pour garantir que le processus d'amplification est toujours en phase exponentielle. Le nombre de cycles d'amplification nécessaires pour atteindre la ‘valeur seuil' est appelé ‘cycle seuil' (Cs en français, Ct en anglais). C'est grâce au suivi en temps réel de l'amplification d'ADN que le cycle seuil peut être déterminé.
Le fait qu'il y ait une valeur seuil unique Qs pour tous les échantillons traités dans une même expérience a pour conséquence que la quantité initiale d'ADN présente dans les différents échantillons analysés en parallèle est directement reliée à la valeur du cycle seuil Cs.
On a alors, pour les échantillons 1,2,3,...,n :
![]()
![]()
![]()
...
![]()
Il est alors très facile de calculer le rapport entre les quantités initiales d'ADN de deux échantillons dont les ‘cycles seuils' sont respectivement Cs1 et Cs2*. Ce rapport est directement dépendant de la différence entre les valeurs des ‘cycles seuils':
|
Efficacité de duplication et détermination expérimentale de cette efficacité
L'efficacité de la duplication de l'ADN est un paramètre crucial pour le calcul des quantités d'ADN amplifiées ou présentes à l'origine.
L'efficacité d'amplification dépend des conditions de la PCR. Dans des conditions "standardisées" (même polymérase et même tampon de réaction), l'efficacité dépend principalement du couple d'amorces utilisé et de la température à laquelle l'hybridation des amorces avec la matrice est réalisée.
Il est souhaitable d'avoir des efficacités de duplication les plus proches de 100% car cela garantit que l'amplification de l'ADN se déroule dans des conditions optimales. Pour atteindre cet objectif, il faut soit optimiser la température d'hybridation des amorces avec la matrice, soit définir de nouvelles amorces mieux adaptées si l'optimisation ne suffit pas.
Il est possible de travailler avec des efficacités inférieures à 100% (par exemple comprises entre 80 et 100%), mais cela doit se faire sous réserve que la valeur d'efficacité soit reproductible d'un échantillon à l'autre.
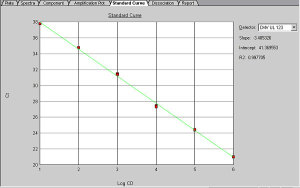
L'efficacité d'un couple d'amorces à dupliquer la matrice d'ADN se détermine de manière expérimentale. Une expérience de PCR quantitative est réalisée en considérant comme échantillons différentes dilutions d'une même matrice. Plus la matrice est diluée, plus la valeur du ‘cycle seuil' sera élevée et on s'attend à une relation linéaire liant le logarithme de la concentration en ADN des différents échantillons de matrice et le cycle seuil (voir figure). En effet, si par exemple on dilue deux fois l'ADN matrice, on s'attend à ce qu'il faille un cycle d'amplification supplémentaire pour atteindre la ‘valeur seuil' de fluorescence si l'efficacité de duplication de l'ADN est de 100%. |
Dans une expérience de PCR quantitative réalisée en considérant comme échantillons différentes dilutions d'une même matrice et en reprenant la formule (4) indiquée ci-dessus Q01 / Q02 = (1+E)(Cs2 – Cs1), le calcul permet d'accéder directement à la valeur d'efficacité. En effet, la formule (4) lie l'efficacité de duplication de l'ADN que l'on cherche à déterminer, avec le rapport entre les quantités initiales en ADN et la différence entre les valeurs de ‘cycle seuil' qui sont tous deux connus. Ainsi, pour deux échantillons dont les quantités initiales en ADN varient d'un facteur 10, si la différence entre les Cs est de 3,5, on obtient, par application de la formule (4) ci-dessus une efficacité de duplication de l'ADN de 93% :
![]() (5)
(5)
![]() (6)
(6)
Réalisation pratique d'une réaction de PCR quantitative
Comme dans le cas de la réalisation d'une PCR normale, le mélange réactionnel nécessaire pour réaliser une expérience de PCR quantitative doit comprendre :
Comme pour la PCR « classique » :
Un composé supplémentaire :
|
Pour toute expérience de PCR quantitative, il est nécessaire de prévoir un certain nombre d'échantillons témoins permettant de mesurer expérimentalement l'efficacité d'amplification, selon la méthode décrite ci-dessus.
Les échantillons d'intérêt sont en général répétés en trois exemplaires, pour limiter les risques d'erreur de mesure.
Il est important de noter que dans la très grande majorité des applications, l'évaluation de la quantité d'ADN est comparative : comparaison des nombres de copies de gènes entre différents individus, mesure de la proportion d'un ingrédient ou d'un contaminant dans un produit, comparaison du niveau d'expression de deux gènes, effet d'un traitement sur l'expression d'un gène...
Pour aborder ces questions, toute expérience de PCR quantitative se doit de prévoir l'analyse d'échantillons contrôles ou références, ces contrôles étant des cibles dont on connaît par ailleurs la quantité ou l'abondance. Pour les études d'expression de gène, le contrôle est par exemple un gène dont l'expression est stable ou indépendante du traitement étudié. Pour les mesures d'abondance d'ingrédients ou de contaminants, on peut se référer à des standards calibrés (voir l'exercice d'application sur la détection d'OGMs).